picrust2功能预测-从qiime2安装到数据分析_picrust2功能预测结果怎么看-程序员宅基地
技术标签: github linux picrust2 windows
记录一下跌跌撞撞地摸索过程
picrust2功能预测
参照公众号《宏基因组》刘永鑫的原创:《PICRUSt2:OTU或ASV等16S随便预测宏基因组且数据库增加10倍》https://blog.csdn.net/woodcorpse/article/details/89302863
注:命令中 \为断行用,实际上输入命令要去掉,在输入文件路径时没有用此符号断行。
虚拟机与qiime2安装
参照公众号《宏基因组》刘永鑫的原创:《QIIME 2用户文档. 01简介和安装 Introduction & Install(2020.2) 》使用VirtualBox方式安装部分
https://blog.csdn.net/woodcorpse/article/details/75103929
下载最新版本虚拟机:https://download.virtualbox.org/virtualbox/6.1.4/VirtualBox-6.1.4-136177-Win.exe
下载最新版本虚拟机扩展包:
https://download.virtualbox.org/virtualbox/6.1.4/Oracle_VM_VirtualBox_Extension_Pack-6.1.4.vbox-extpack
下载QIIME2镜像:https://data.qiime2.org/distro/core/2020.2
安装:
1) 安装最新版虚拟机,默认位置,装好后将扩展包拷贝到安装目录windows/program files/oracle/virtualbox,双击安装扩展包。
2) 双击下载好的QIIME2镜像或在虚拟机界面点击导入镜像,修改分配给虚拟机的cpu和内存(一般是windows的一半),点击导入。
3) 启动虚拟机,菜单中安装Guest Additions,按照提示完成后,重启linux系统才能设置共享文件夹和共享复制粘贴。https://mp.weixin.qq.com/s/WS9u0nhiS1eizL5KXKs__A?
4) 设置共享文件夹,添加共享文件夹,勾选固定分配/自动挂载。
问题1:安装好后,输入qiime的命令均显示command not found。
前几天看到有人在百度上发起了同样问题。不是安装有问题,是qiime的命令和qiime2的命令不兼容,因此使用qiime2进行16分析的命令可参照https://www.cnblogs.com/afeiyuanda/p/11037287.html
Picrust2安装
https://github.com/picrust/picrust2/官网有给出两种安装方法,建议第一种安装,不会出错。
安装好后第二次使用picrust,也要输入激活命令:
conda activate picrust2
或者
conda activate picrust
如果想要使用qiime2命令,则要先退出picrust2,输入失活命令:
conda deactivate
问题2:v2.3.2-b版本优化了匹配度,测序序列进行后续分析时都会出现这样的问题,stopping - all 234 input sequences aligned poorly to reference sequence(--min_align option specified a minimum proportion of 0.8 aligning to reference sequences
先反向互补完,再做功能预测就可以了,seqtk seq -r otus.fa.format.fasta > otus_rc.fa
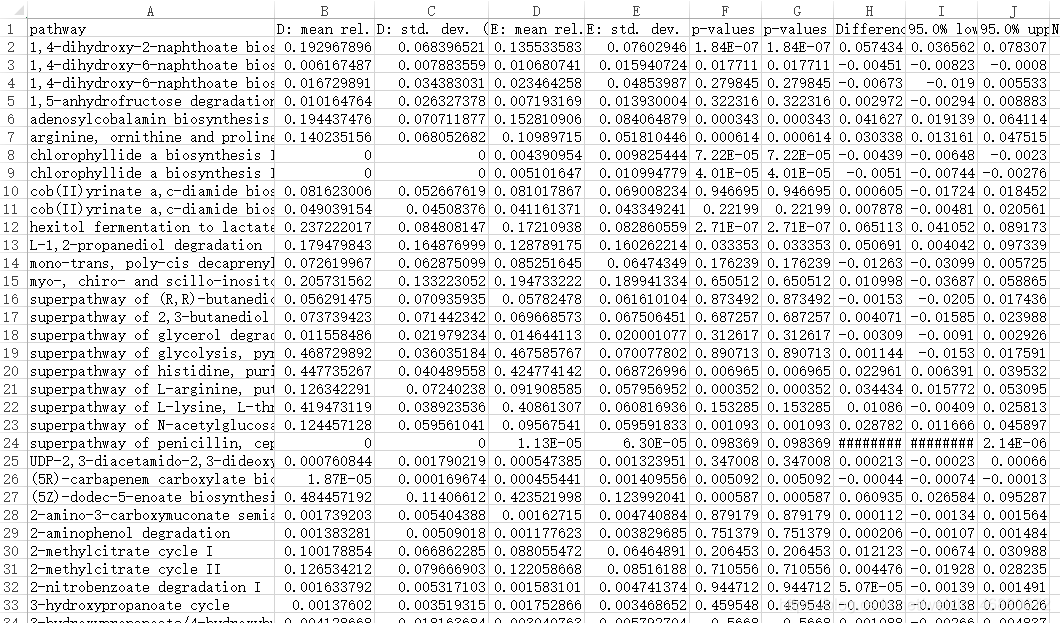
如果使用以前的版本不会出现这样的问题。
有个小插曲:不知道怎么解决这个问题的时候,无论输入什么数据都显示出这个结果,心态差点就崩了,然后花了点钱请淘宝上做生信分析的人,用我的数据帮我跑了一遍,可以出结果。最后观察版本之间有什么差别的时候就发现最新版本优化了,然后淘宝上那个生信技术厉害的客服用最新版本也出不了结果,他就去官网发了一个issue(就是上面图片里的),开发者很快就给回复了,解释了原因。
问题3:使用vsearch跑出的结果使用out.fa和out_table.biom两个文件可以直接跑出预测结果,但使用qiime跑出的结果中两个文件夹的out名称不一致,要删掉后面的其他字符可采用以下方法。
修改fastq文件OTU 名字`
awk '{print $1 }' FASTA_IN > FASTA_OUT
其中FASTA_IN和FASTA_OUT是输入和输出FASTA文件的名称。
$2:一行一行的读取指定的文件, 以空格作为分隔符,打印第二个字段
比如有这样一个文件
a1 b1 c1 d1
a2 b2 c2 d2
执行的结果是,输出:
b1
b2
问题4:会出现以下报错,大概率是电脑内存不够,picrust2官网有一个针对这个错误的issue。
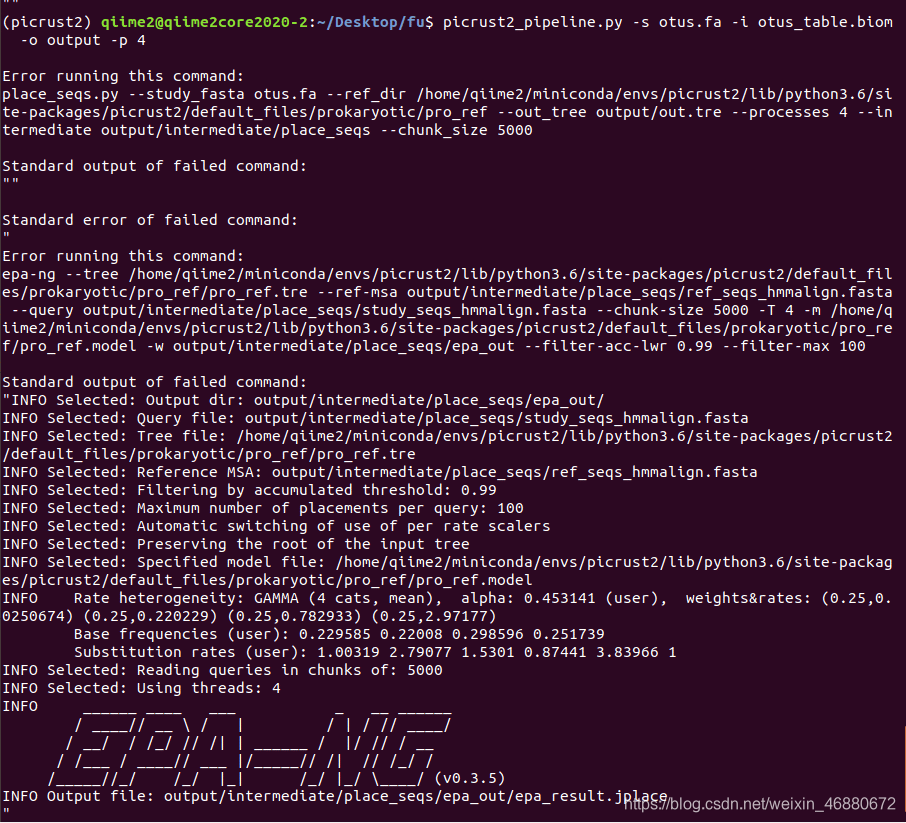
解决的根本办法是扩大你的内存,建议32G往上走,windows32G内存一般能够给虚拟机分配20G左右运行内存,在样本量比较多的情况下,往往16G是不够的,但是有时候样本量可以的情况下可以打擦边球试运行,降低CPU线程。我之前运行过分配给虚拟机36G的内存(windows总共48G)运行104个样本,使用4和2线程均无法得到结果,之后再使用1个线程耗时19000秒就成功了。也有试过88个样本,2线程9000秒。
###picrust2预测
比较完整的一步脚本是
picrust2_pipeline.py -s dna-sequences.fasta -i feature-table.biom -o \
picrust2_out_pipeline -p 20 -r default_files/prokaryotic/pro_ref/pro_ref \
--in_traits COG,EC,KO,PFAM,TIGRFAM
可能default_files/prokaryotic/pro_ref/pro_ref这个文件需要你在你的虚拟机上搜索后输入完整并且正确的文件路径。
这个命令运行后的结果包括COG,EC,KO,PFAM,TIGRFAM的丰度信息和MetaCyc途径丰度。
我一般输入的命令是:
picrust2_pipeline.py –s out.fa -i out_table.biom \
-o picrust2_out_pipeline -p 4
-p 4是使用4线程的意思
输出的结果包括EC,KO,PATHWAY的丰度信息。
但是但是结果文件中的结果比较简洁,比如只输入EC1.1.1,而不标注对应的酶名字,这时候需要加工加工。根据需要酌情添加描述:
add_descriptions.py -i EC_metagenome_out/pred_metagenome_unstrat.tsv \
-m EC -o EC_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i KO_metagenome_out/pred_metagenome_unstrat.tsv \
-m KO -o KO_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i COG_metagenome_out/pred_metagenome_unstrat.tsv \
-m COG -o COG_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i PFAM_metagenome_out/pred_metagenome_unstrat.tsv \
-m PFAM -o PFAM_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i TIGRFAM_metagenome_out/pred_metagenome_unstrat.tsv\
-m TIGRFAM -o TIGRFAM_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i pathways_out/path_abun_unstrat.tsv
-m METACYC -o pathways_out/path_abun_unstrat_descrip.tsv
结果输出中没有KEGG结果,可通过以下命令生成KEGG pathway 丰度表和注释结果
pathway_pipeline.py -i KO_metagenome_out/pred_metagenome_unstrat.tsv \
-o KEGG_pathways_out --no_regroup --map \
/home/qiime2/miniconda/envs/picrust/lib/python3.6/site-package/
picrust2/default_files/pathway_mapfiles/KEGG_pathways_to_KO.tsv
这里KEGG_pathways_to_KO.tsv需要你在你的虚拟机上搜索后输入完整并且正确的文件路径。
# 添加功能描述
add_descriptions.py -i KEGG_pathways_out/path_abun_unstrat.tsv.gz \
--custom_map_table /home/qiime2/miniconda/envs/picrust/lib/python3.6/site-
package/picrust2/default_files/description_mapfiles/KEGG_pathways_info.tsv.gz \
-o KEGG_pathways_out/path_abun_unstrat_descrip.tsv.gz
这里/KEGG_pathways_info.tsv.gz需要你在你的虚拟机上搜索后输入完整并且正确的文件路径。
如果不懂可看原文https://www.jianshu.com/p/e3beaee77423
或如果不想一步生成结果,就看官网分步命令:https://github.com/picrust/picrust2/wiki/PICRUSt2-Tutorial-(v2.1.4-beta)
分析好的结果,使用STAMP软件进行显著性差异分析,该软件导入相应数据后既可以直接出很直观漂亮的显著性差异图,也可一次性导出所有通路或酶的组间差异分析详细数据。如下两图所示。
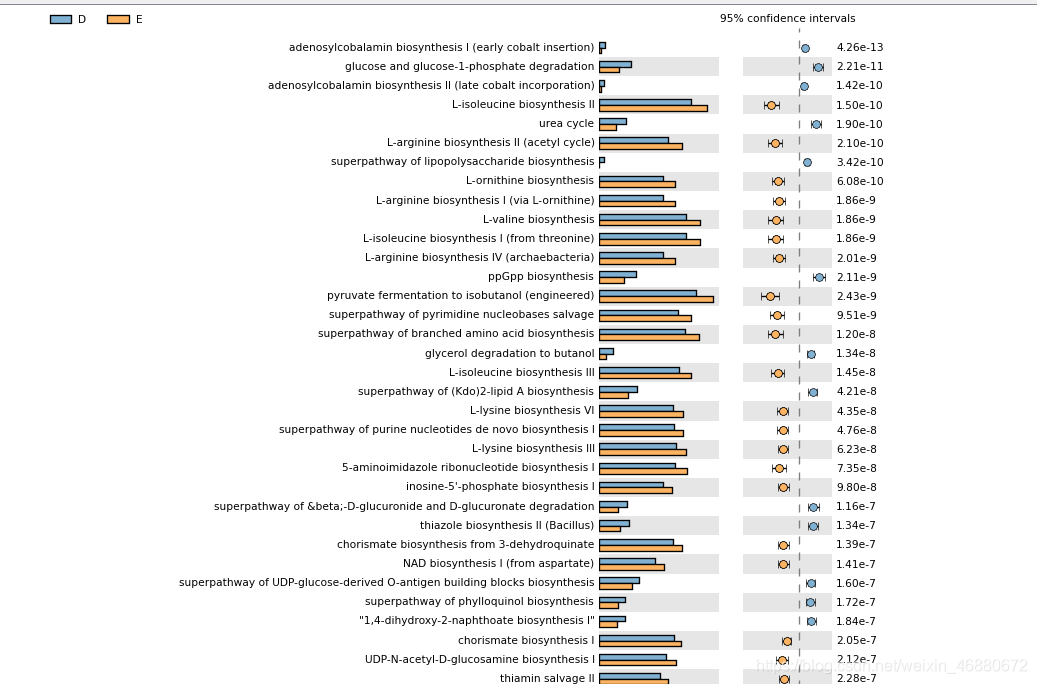
智能推荐
c# 调用c++ lib静态库_c#调用lib-程序员宅基地
文章浏览阅读2w次,点赞7次,收藏51次。四个步骤1.创建C++ Win32项目动态库dll 2.在Win32项目动态库中添加 外部依赖项 lib头文件和lib库3.导出C接口4.c#调用c++动态库开始你的表演...①创建一个空白的解决方案,在解决方案中添加 Visual C++ , Win32 项目空白解决方案的创建:添加Visual C++ , Win32 项目这......_c#调用lib
deepin/ubuntu安装苹方字体-程序员宅基地
文章浏览阅读4.6k次。苹方字体是苹果系统上的黑体,挺好看的。注重颜值的网站都会使用,例如知乎:font-family: -apple-system, BlinkMacSystemFont, Helvetica Neue, PingFang SC, Microsoft YaHei, Source Han Sans SC, Noto Sans CJK SC, W..._ubuntu pingfang
html表单常见操作汇总_html表单的处理程序有那些-程序员宅基地
文章浏览阅读159次。表单表单概述表单标签表单域按钮控件demo表单标签表单标签基本语法结构<form action="处理数据程序的url地址“ method=”get|post“ name="表单名称”></form><!--action,当提交表单时,向何处发送表单中的数据,地址可以是相对地址也可以是绝对地址--><!--method将表单中的数据传送给服务器处理,get方式直接显示在url地址中,数据可以被缓存,且长度有限制;而post方式数据隐藏传输,_html表单的处理程序有那些
PHP设置谷歌验证器(Google Authenticator)实现操作二步验证_php otp 验证器-程序员宅基地
文章浏览阅读1.2k次。使用说明:开启Google的登陆二步验证(即Google Authenticator服务)后用户登陆时需要输入额外由手机客户端生成的一次性密码。实现Google Authenticator功能需要服务器端和客户端的支持。服务器端负责密钥的生成、验证一次性密码是否正确。客户端记录密钥后生成一次性密码。下载谷歌验证类库文件放到项目合适位置(我这边放在项目Vender下面)https://github.com/PHPGangsta/GoogleAuthenticatorPHP代码示例://引入谷_php otp 验证器
【Python】matplotlib.plot画图横坐标混乱及间隔处理_matplotlib更改横轴间距-程序员宅基地
文章浏览阅读4.3k次,点赞5次,收藏11次。matplotlib.plot画图横坐标混乱及间隔处理_matplotlib更改横轴间距
docker — 容器存储_docker 保存容器-程序员宅基地
文章浏览阅读2.2k次。①Storage driver 处理各镜像层及容器层的处理细节,实现了多层数据的堆叠,为用户 提供了多层数据合并后的统一视图②所有 Storage driver 都使用可堆叠图像层和写时复制(CoW)策略③docker info 命令可查看当系统上的 storage driver主要用于测试目的,不建议用于生成环境。_docker 保存容器
随便推点
网络拓扑结构_网络拓扑csdn-程序员宅基地
文章浏览阅读834次,点赞27次,收藏13次。网络拓扑结构是指计算机网络中各组件(如计算机、服务器、打印机、路由器、交换机等设备)及其连接线路在物理布局或逻辑构型上的排列形式。这种布局不仅描述了设备间的实际物理连接方式,也决定了数据在网络中流动的路径和方式。不同的网络拓扑结构影响着网络的性能、可靠性、可扩展性及管理维护的难易程度。_网络拓扑csdn
JS重写Date函数,兼容IOS系统_date.prototype 将所有 ios-程序员宅基地
文章浏览阅读1.8k次,点赞5次,收藏8次。IOS系统Date的坑要创建一个指定时间的new Date对象时,通常的做法是:new Date("2020-09-21 11:11:00")这行代码在 PC 端和安卓端都是正常的,而在 iOS 端则会提示 Invalid Date 无效日期。在IOS年月日中间的横岗许换成斜杠,也就是new Date("2020/09/21 11:11:00")通常为了兼容IOS的这个坑,需要做一些额外的特殊处理,笔者在开发的时候经常会忘了兼容IOS系统。所以就想试着重写Date函数,一劳永逸,避免每次ne_date.prototype 将所有 ios
如何将EXCEL表导入plsql数据库中-程序员宅基地
文章浏览阅读5.3k次。方法一:用PLSQL Developer工具。 1 在PLSQL Developer的sql window里输入select * from test for update; 2 按F8执行 3 打开锁, 再按一下加号. 鼠标点到第一列的列头,使全列成选中状态,然后粘贴,最后commit提交即可。(前提..._excel导入pl/sql
Git常用命令速查手册-程序员宅基地
文章浏览阅读83次。Git常用命令速查手册1、初始化仓库git init2、将文件添加到仓库git add 文件名 # 将工作区的某个文件添加到暂存区 git add -u # 添加所有被tracked文件中被修改或删除的文件信息到暂存区,不处理untracked的文件git add -A # 添加所有被tracked文件中被修改或删除的文件信息到暂存区,包括untracked的文件...
分享119个ASP.NET源码总有一个是你想要的_千博二手车源码v2023 build 1120-程序员宅基地
文章浏览阅读202次。分享119个ASP.NET源码总有一个是你想要的_千博二手车源码v2023 build 1120
【C++缺省函数】 空类默认产生的6个类成员函数_空类默认产生哪些类成员函数-程序员宅基地
文章浏览阅读1.8k次。版权声明:转载请注明出处 http://blog.csdn.net/irean_lau。目录(?)[+]1、缺省构造函数。2、缺省拷贝构造函数。3、 缺省析构函数。4、缺省赋值运算符。5、缺省取址运算符。6、 缺省取址运算符 const。[cpp] view plain copy_空类默认产生哪些类成员函数